pandas高效读取大文件的探索之路
使用
pandas
进行数据分析时,第一步就是读取文件。
在平时学习和练习的过程中,用到的数据量不会太大,所以读取文件的步骤往往会被我们忽视。
然而,在实际场景中,面对十万,百万级别的数据量是家常便饭,即使千万,上亿级别的数据,单机处理也问题不大。
不过,当数据量和数据属性多了之后,读取文件的性能瓶颈就开始浮现出来。
当我们第一次拿到数据时,经常会反反复复的读取文件,尝试各种分析数据的方法。
如果每次读取文件都要等一段时间,不仅会影响工作效率,还影响心情。
下面记录了我自己优化
pandas
读取大文件效率的探索过程。
1. 准备部分
首先,准备数据。
下面的测试用的数据是一些虚拟币的交易数据,除了常用的K线数据之外,还包含很多分析因子的值。
import pandas as pd
fp = "all_coin_factor_data_12H.csv"
df = pd.read_csv(fp, encoding="gbk")
df.shape
# 运行结果
(398070, 224)
总数据量接近
40万
,每条数据有
224
个属性。
然后,封装一个简单的
装饰器
来计时函数运行时间。
from time import time
def timeit(func):
def func_wrapper(*args, **kwargs):
start = time()
ret = func(*args, **kwargs)
end = time()
spend = end - start
print("{} cost time: {:.3f} s".format(func.__name__, spend))
return ret
return func_wrapper
2. 正常读取
先看看读取这样规模的数据,需要多少时间。
下面的示例中,循环读取
10次
上面准备的数据
all_coin_factor_data_12H.csv
。
import pandas as pd
@timeit
def read(fp):
df = pd.read_csv(
fp,
encoding="gbk",
parse_dates=["time"],
)
return df
if __name__ == "__main__":
fp = "./all_coin_factor_data_12H.csv"
for i in range(10):
read(fp)
运行结果如下:
读取一次大概
27秒
左右。
3. 压缩读取
读取的文件
all_coin_factor_data_12H.csv
大概
1.5GB
左右,
pandas
是可以直接读取压缩文件的,尝试压缩之后读取性能是否能够提高。
压缩之后,大约
615MB
左右,压缩前大小的一半不到点。
import pandas as pd
@timeit
def read_zip(fp):
df = pd.read_csv(
fp,
encoding="gbk",
parse_dates=["time"],
compression="zip",
)
return df
if __name__ == "__main__":
fp = "./all_coin_factor_data_12H.zip"
for i in range(10):
read_zip(fp)
运行结果如下: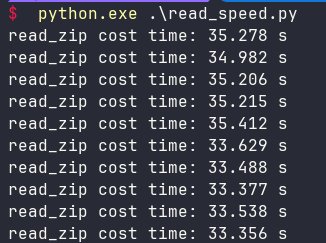
读取一次大概
34秒
左右,还不如直接读取来得快。
4. 分批读取
接下来试试分批读取能不能提高速度,分批读取的方式是针对数据量特别大的情况,
单机处理过亿数据量的时候,经常会用到这个方法,防止内存溢出。
先试试每次读取
1万条
:
import pandas as pd
@timeit
def read_chunk(fp, chunksize=1000):
df = pd.DataFrame()
reader = pd.read_csv(
fp,
encoding="gbk",
parse_dates=["time"],
chunksize=chunksize,
)
for chunk in reader:
df = pd.concat([df, chunk])
df = df.reset_index()
return df
if __name__ == "__main__":
fp = "./all_coin_factor_data_12H.csv"
for i in range(10):
read_chunk(fp, 10000)
运行结果如下: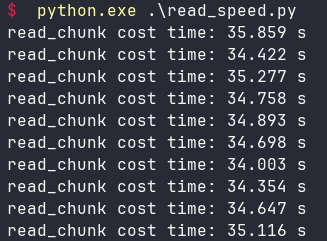
和读取压缩文件的性能差不多。
如果调整成每次读取
10万条
,性能会有一些微提高。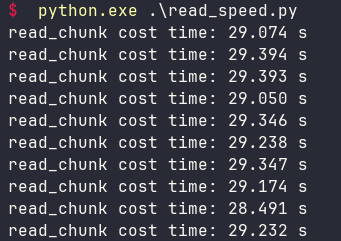
分批读取时,一次读取的越多(只要内存够用),速度越快。
其实我也试了一次读取
1千条
的性能,非常慢,这里就不截图了。
5. 使用polars读取
前面尝试的方法,效果都不太好,下面引入一个和
pandas
兼容的库
Polars
。
Polars
是一个高性能的
DataFrame
库,它主要用于操作结构化数据。
它是用
Rust
写的,主打就是
高性能
。
使用
Polars
读取文件之后返回的
Dataframe
虽然和
pandas
的
DataFrame
不完全一样,
当可以通过一个简单的
to_pandas
方法来完成转换。
下面看看使用
Polars
读取文件的性能:
import polars as pl
@timeit
def read_pl(fp):
df = pl.read_csv(
fp,
encoding="gbk",
try_parse_dates=True,
)
return df.to_pandas()
if __name__ == "__main__":
fp = "./all_coin_factor_data_12H.csv"
for i in range(10):
read_pl(fp)
运行结果如下: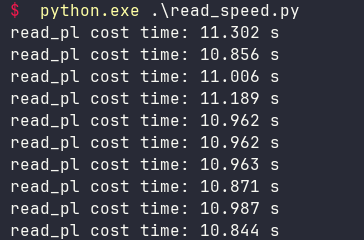
使用
Polars
后性能提高非常明显,看来,混合使用
Polars
和
pandas
是一个不错的方案。
6. 序列化后读取
最后这个方法,其实不是直接读取原始数据,而是将原始数据转换为
python
自己的
序列化格式
(
pickle
)之后,再去读取。
这个方法多了一个转换的步骤:
fp = "./all_coin_factor_data_12H.csv"
df = read(fp)
df.to_pickle("./all_coin_factor_data_12H.pkl")
生成一个 序列化文件:
all_coin_factor_data_12H.pkl
。
然后,测试下读取这个序列化文件的性能。
@timeit
def read_pkl(fp):
df = pd.read_pickle(fp)
return df
if __name__ == "__main__":
fp = "./all_coin_factor_data_12H.pkl"
for i in range(10):
read_pkl(fp)
运行结果如下: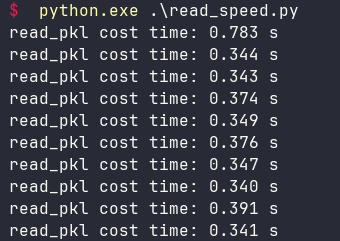
这个性能出乎意料之外的好,而且
csv文件
序列化成
pkl文件
之后,占用磁盘的大小也只有原来的一半。
csv
文件
1.5GB
左右,
pkl
文件只有
690MB
。
这个方案虽然性能惊人,但也有一些局限,
首先是原始文件不能是那种实时变化的数据,因为原始
csv文件
转换为
pkl文件
也是要花时间的(上面的测试没有算这个时间)。
其次,序列化之后的
pkl文件
是
python
专用的,不像
csv文件
那样通用,不利于其他
非python
的系统使用。
7. 总结
本文探讨了一些
pandas
读取大文件的优化方案,最后比较好的就是
Polars方案
和
pickle序列化
方案。
如果我们的项目是分析固定的数据,比如历史的交易数据,历史天气数据,历史销售数据等等,
那么,就可以考虑
pickle序列化
方案,先花时间讲原始数据序列化,
后续的分析中不担心读取文件浪费时间,可以更高效的尝试各种分析思路。
除此之外的情况,建议使用
Polars方案
。
最后补充一点,如果读取文件的性能对你影响不大,那就用原来的方式,千万不要画蛇添足的去优化,
把精力花在数据分析的业务上。